Nortable Research
Current challenges and best practices
for cell-free long RNA biomarker
discovery
Technology Trend
학회뉴스
연세의대
지난 몇 년 동안, 질병 특이 생체 표지자(biomarker)를 검출하기 위한 비침습적인 방법이 등장하고 있습니다. 특히 액체 생검 방법에 대한 연구가 활발히 진행되었는데, 이 중 대부분은 DNA 기반의 연구입니다. 이에 관련 된 리뷰 논문으로 Long RNA 기반의 액체 생검의 적용에 대해 소개하고자 합니다.
현재 대부분의 순환 DNA 연구는 종양 DNA (ctDNA)에 중점을 두고 있으며, 이는 종양에서 방출된 DNA 분자로 특정 돌연변이 정보를 제공합니다. 그러나 ctDNA의 풍부도는 종양 부담(Tumor burden)과 직접적으로 관련되어 암의 조기 검출에 제한이 있습니다. 이와 달리 세포 외 RNA (cell-free RNA, cfRNA)은 암이 아닌 세포에서도 분비되어 RNA 발현의 변화를 동적으로 반영합니다. 또한 cfRNA의 연구는 DNA에서 확인할 수 없는 특정 유전자군의 발현성 차이를 비롯해 병원성 대체 스플라이싱이나 A-to-I RNA 편집(A-to-I RNA editing) 등도 확인할 수 있습니다.
cfRNA 연구는 주로 혈중에서의 마이크로RNA (miRNA)에 중점을 두었지만, 최근에는 메신저 RNA (mRNA) 및 긴 비코딩 RNA (lncRNA)를 포함한 long cfRNA에 대한 연구가 증가하고 있습니다.혈액에서 cfRNA을 검출하는 기술적 어려움은 있지만, long RNA의 연구는 알려진 miRNA보다 수가 훨씬 많아서 특정 질병의 상태를 높은 신뢰성으로 식별하는 데 잠재력이 큽니다. cfRNA의 검출 과정은 다음과 같습니다 (그림 1).검사 과정 시 유의해야 할 점은 다음과 같습니다 (그림 2).
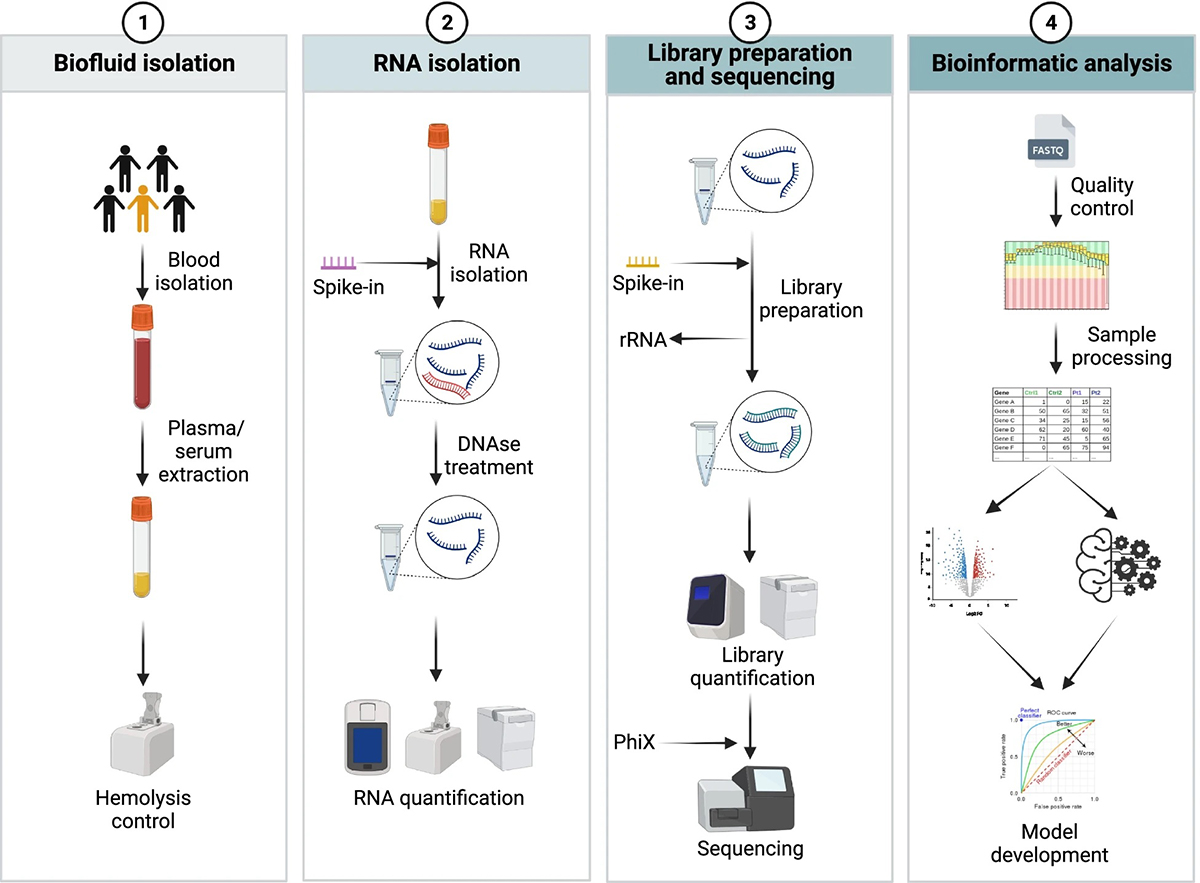 <그림 1> Schematic timeline of all the steps involved in the development of cfRNA biomarkers
<그림 1> Schematic timeline of all the steps involved in the development of cfRNA biomarkers
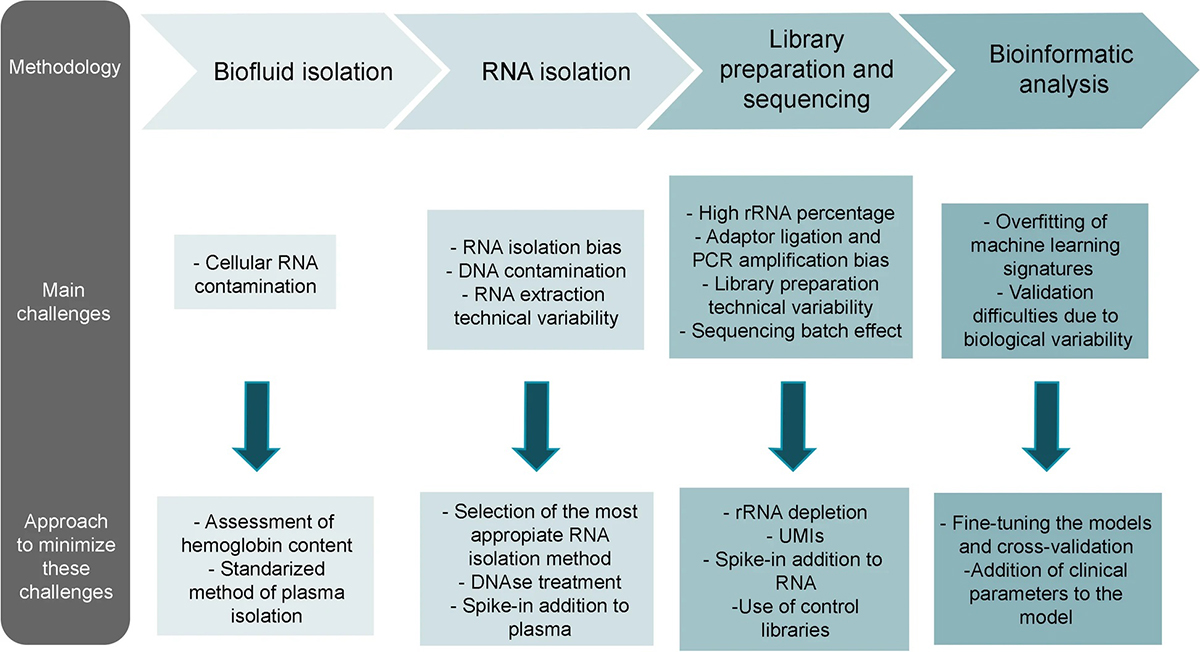 <그림 2> Overview of the main challenges in the process of biomarker discovery and specific steps to minimize them
<그림 2> Overview of the main challenges in the process of biomarker discovery and specific steps to minimize them
Biofluid isolation
혈액에서의 cfRNA은 미세 소포나 리보핵단백질 복합체에 캡슐화되어 있습니다. 혈액 세포로부터의 RNA 오염을 줄이기 위해 혈청 또는 혈장이 검체로 주로 사용됩니다. 두 검체 종류 간 결과 차이 여부는 아직 논란이 있으나 혈액 응고
중에 cfRNA이 혈전에 부착될 수 있어 혈장에 비해 혈청의 cfRNA의 양이 감소할 수 있다는 보고가 있습니다. 검체 처리 중 혈소판, 혈소판 유래 RNA, 용혈 과정의 RBC RNA, 동결-해동 과정 및 검체 보관 기간은 cfRNA 검사에 영향
을 미칠 수 있는 중요한 요인입니다. 세포 유래 오염을 피하고 평가하기 위해 정확한 처리와 품질 관리가 필요하며, 동결-해동 주기와 장기 저장을 최소화하여 추출된 RNA의 품질을 향상시켜 더 견고하고 재현 가능한 결과를 얻을 수 있습니다.
RNA isolation
RNA 분리는 다양한 방법이 존재하며, 이러한 방법은 회수율과 선택적인 RNA 유형의 농축에 영향을 미칩니다. 전통적인 guanidium-thiocyanate나 페놀-클로로포름 방법 대신에 현재는 컬럼 기반 키트가 더 널리 사용되고 있습니다. 그러나 키트의 사용은 RNA 분리 수획율과 같은 내재적인 기술적 차이로 인한 편향(bias)이 발생할 수 있기 때문에, 이러한 편향을 최소화하는 것이 중요합니다. 이를 위한 전략으로는 추출 키트의 일관성을 유지하는 것이 필요합니다. 이를 위해 endogenous RNA와 유사한 GC content를 가진 exogenous RNA를 스파이크-인 (spike-in)하여 RNA 추출 효율을 확인하고, 낮은 RNA 품질을 가진 샘플을 평가하며, 추가적으로 DNAse로 처리하여 DNA 오염을 제거하는 등의 방법을 적용할 수 있습니다.
Library preparation and sequencing
RNA 시퀀싱(RNA-seq)은 생체 표지자 발견의 표준 기법으로 인식되며, 최근 10년 동안 큰 발전을 거쳐 샘플 내에서 알려진 및 새로운 전사체를 탐지하고 양적으로 측정할
수 있게 되었습니다. 이 기술은 다른 방법보다 더 높은 감도와 정확성을 제공합니다. 기술 발전과 가격 하락으로 NGS가 생체 표지자 발견과 스크리닝 도구로 활용되기 시작했으며, 적은 양의 RNA로도 RNA-seq을 수행하기 위한 다양한 라이브러리 준비 프로토콜이 개발되었습니다.
cfRNA 검사를 위한 라이브러리 준비 과정에서는 세포 내 RNA의 대다수인 rRNA를 제거하여 cfRNA를 선별하는 것이 필요합니다. cfRNA는 polyA tail이 존재하지 않기 때문에, polyA enrichment 대신 rRNA 제거 방법을 사용합니다. 이러한 접근 방식은 cfRNA를 효과적으로 분리하고 실험 결과의 정확성을 높이는 데 도움이 됩니다.
라이브러리 준비 과정에서는 어댑터 연결 및 PCR 증폭에 따른 편향도 문제가 발생합니다. 이는 일부 서열이 다른 서열보다 더 쉽게 어댑터 서열에 연결되고 증폭되는 경향이 있기 때문입니다. 이러한 편향성을 줄이기 위해 많은 프로토콜에서는 Unique Molecular Identifiers (UMIs)를 사용하도록 도입되었습니다. UMIs는 PCR 증폭 이전에 DNA/RNA 분자에 연결되는 무작위 서열입니다. UMIs의 사용은 동일한 초기 분자에서 유래한 PCR-duplicated read를 식별하고 이 클론성 증폭을 가상으로 보정하는 데 도움을 줍니다. 특히 UMIs가 어댑터 연결 이전에 적용되면 어댑터 연결 편향성을 최소화하는 데 도움이 됩니다.
UMIs 의 사용은 cfRNA 샘플과 같이 매우 적은 양에서 분화된 유전자 발현 분석의 재현성을 향상시키는 데 기여합니다.
라이브러리 준비 단계에서 유용한 기술 중 하나는 “스파이크-인 (spike-in)”입니다. 스파이크-인은 미리 알려진 농도로 추가되어 라이브러리 준비 중의 증폭 편향을 보정하는 데 활용됩니다. 이는 측정된 서열의 정량화에 도움이 됩니다. 뿐만 아니라, 라이브러리 시퀀싱 과정에서 종종 발생하는 batch effect를 완화하기 위해 상용화된 대조(control) 라이브러리를 도입하한다면 시퀀싱의 재현성을 측정하고 batch effect를 보정할 수 있습니다.
Bioinformatics analysis
RNA-seq를 활용한 생물표지자 발견은 데이터의 컴퓨터
분석이 핵심 단계 중 하나입니다. 생물정보학 분석은 데이터 품질 관리부터 전사량 측정, 다양한 다운스트림 분석까지 여러 단계를 거칩니다. 분석 결과는 데이터 입력 품질과 밀접한 관련이 있기 때문에 품질 평가는 중요한 단계입니다. 가장 흔한 문제 중 하나는 RNA-seq 데이터 처리에서 기술적 편향을 보정하는 과정으로 스파이크인 컨트롤 또는 라이브러리 크기나 유전자 길이에 따른 정규화 (normal-ization) 방법이 있습니다. 스파이크인 컨트롤을 이용한 정규화 방법은 정량적으로 유전자 발현을 확인할 수 있다는 장점이 있으나 스파이크인 컨트롤의 변동성이 큰 증폭으로 인한 문제가 제기되었습니다. UMI 중복 제거 방법 역시 재현성을 향상시킬 수 있는 방법 중 하나입니다.
데이터 처리 후에는 cfRNA 프로파일의 비교 분석 및 기계 학습(machine learning, ML)을 통해 가능성 있는 bio-marker를 찾을 수 있습니다. 표현형과 연관된 유전자를 찾는 전략과 ML 알고리즘이 선택한 여러 유전자를 결합하는 방식은 각각 장단점이 있습니다. ML 방식이 일반적으로 높은 감도와 특이도를 가지는 경향이 있지만, 상대적으로 작은 데이터셋에서는 과적합 (overfitting) 문제에 주의해야 합니다. 교차 검증과 모델 단순화는 ML 결과의 신뢰성을 높이는데 도움이 되는 전략 중 하나입니다. 이러한 방법은 이미 다양한 질병의 진단이나 예후 예측에 성공적으로 적용된 바 있습니다. 또한, 다양한 연구 그룹들은 새로운 방법론을 제안하여 결과의 재현성을 향상시키고 특이적인biomarker를 찾는데 기여하고 있습니다. 예를 들어, 정상
(non-cancer) 검체에서 발현되지 않고 암 검체에서만 발현되는 유전자를 이용하여 특이성을 향상시키거나 특정 세포 유형의 전사체 지문 (transcriptomic fingerprint)을 식별하여 cfRNA의 유래 세포 유형을 해독하는 방법론이 제시되었습니다. 이 접근 방식을 사용하면 관심 있는 장기에서 유래한 유전자 집합에만 초점을 맞추어 검사의 재현성을 향상시키고 변동성을 줄일 수 있습니다.
cfRNA 프로파일의 비교 분석은 더 복잡한 ML 시그니처(signature)에 비해 비용이 적게 들어가고 실용성이 향상되어, 임상 실무에 더 적합합니다. 그러나 ML 시그니처는 더 높은 정확도를 가진 경향이 있습니다. 양쪽 방법은 모두 진단과 예후에 대한 높은 가치의 시그니처를 얻는 데 기여하므로, 연구 목적과 자원 상황을 고려하여 적합한 방법을 선택하는 것이 중요합니다.
Limitations of cfRNA biomarker discovery
액체 생검 방법의 임상적 적용의 가장 큰 이슈는 검사의 기술적 및 생물학적 재현성 편향입니다. 이는 종종 샘플 처리 및 데이터 분석을 위한 표준 방법의 부재와 관련이 있습니다. 이 재현성 부족은 이 분야가 직면한 주요 문제 중 하나로, 많은 RNA 시그니처 (RNA signature)가 임상 시험에 진입했지만 진료에 적용되지 못한 큰 요인 중 하나입니다. 이러한 표준화 부재를 완화하기 위해 데이터 저장소 구현이나 참조 RNA 스파이크인 컨트롤 생성과 같은 방법이 제시되었습니다.
샘플 처리 중 도입된 기술적 편향 외에도, 개인의 나이나 성별과 같은 외부 요인은 cfRNA profile에 강력한 영향을 미칩니다. 또한 인간 간의 가변성이 매우 높기 때문에 이러한 편향을 제어하기 위해 연구의 모든 단계는 잘 조절되고 문서화되어야 하며, 모든 요인에서 균형 잡힌 코호트가 있어야 합니다. 균형 잡힌 코호트를 갖기 어려운 경우, 이러한 외부 요인은 통계 분석에서 고려되어야 합니다.
Current challenges in the field of RNA based liquid biopsies
최근의 시장 조사에 따르면, 2026년까지 액체 생검 산업은 58억 달러를 초과할 것으로 예상됩니다. 이미 일부 cfDNA 검사는 임상 진료에서 사용되고 있습니다. 그러나 cfRNA 분야는 아직 초기 단계이며, 진료에 사용되는 검사는 아직 없습니다. 몇몇 연구가 현재 임상 시험을 진행 중이지만, 모든 가능한 편향을 평가하기 위한 견고하고 표준화된 방법론이 필요합니다. 액체 생검이 생체 표지자, 특히 초기 진단을 위한 표지자 검사로 나아가려면 대규모 인구 스크리닝을 포함한 전향적 연구가 필요합니다. 또한, 다양한 연구 단체의 데이터를 통합하여 데이터베이스를 구축하는 것도 필요하나, 이를 위해서는 표준화된 검사 방법이 필요합니다.
Conclusion
액체 생검 분야, 특히 long cfRNA검사는 유망하지만 상대적으로 적은 수의 연구가 발표되어 있으며 현재까지 승인된 생체표지자는 없습니다. 지난 몇 년 동안 miRNA에서 long cfRNA으로 초점이 옮겨가기 시작하면서 새로운 질병과 연관된 더 많은 RNA의 발견이 이루어졌습니다. long cfRNA을 임상 진료에 적용하기까지 많은 작업이 남아 있지만, 최근의 결과들은 long cfRNA 기반 액체 생검이 스크리닝과 진단 분야에서 다음 큰 혁명 중 하나가 될 수 있다는 가능성을 시사합니다.
[References]
Cabús, L., Lagarde, J., Curado, J. et al. Current challenges and best practices for cell-free long RNA biomarker
discovery. Biomark Res 10, 62 (2022). https://doi.org/10.1186/s40364-022-00409-w

